不要な細胞を効率よく除去できる技術が求められている
京都大学は9月3日、がんや自己免疫疾患を起こす細胞など、体内における不要細胞を標的として貪食により除去する新しいタンパク質を開発したと発表した。この研究は、同大アイセムス(高等研究院 物質―細胞統合システム拠点:WPI-iCeMS)の鈴木淳教授、大和勇輝元研究員らの研究グループによるもの。研究成果は、「Nature Biomedical Engineering」に掲載されている。
画像はリリースより
ヒトの体では日々、不要となった細胞がアポトーシス(細胞死)を経て、貪食細胞によって排除されている。しかし、これらの不要細胞が加齢とともに細胞死を起こすことができなくなると、体内に蓄積し、がん、自己免疫疾患、臓器機能の破綻などさまざまな疾患が引き起こされる。
不要細胞に対して細胞死を誘導するために、化学物質や抗体、細胞を用いた治療が行われ、一定の成果をあげてきた。一方で、これらの不要細胞は簡単には細胞死を起こさないため、貪食細胞により直接除去することができれば、より効果的な治療ができる可能性がある。
従来とは異なるアプローチで不要細胞の除去に挑戦
これまでに研究グループは、死細胞において「eat me」シグナルであるホスファチジルセリンを細胞表面に露出させるスクランブラーゼを同定し、研究分野をリードしてきた。また、スクランブラーゼを薬剤などで活性化することで「eat me」シグナルを露出させ、不要細胞の除去を行うアプローチを考案してきた。しかし、研究を進める中で、不要細胞に特異的な細胞表面の変化を認識させることで、「eat me」シグナルの露出がなくても貪食により標的細胞を除去できるのではないかとの考えに至った。
そこで今回の研究では、不要な細胞を安全かつ選択的に排除するための合成タンパク質の開発に取り組んだ。
細胞死を介さず、貪食を誘導する人工タンパク質「クランチ」開発
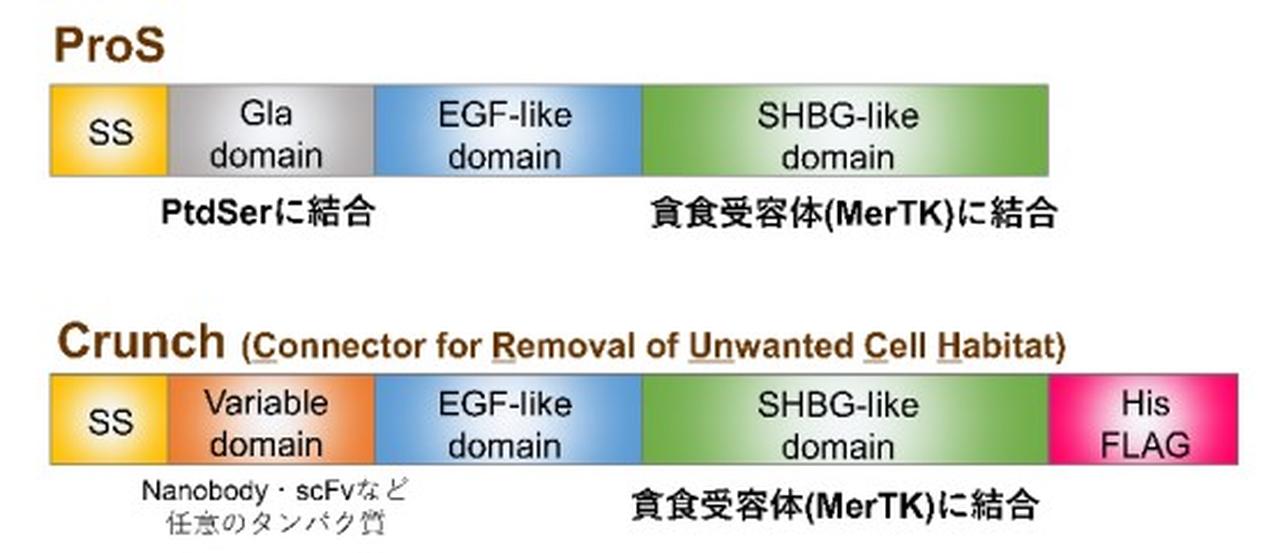
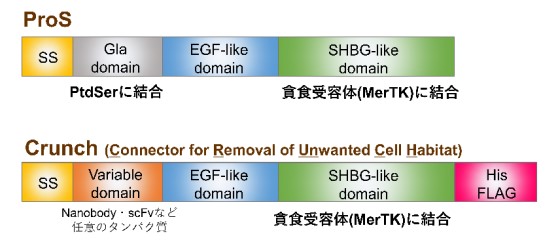
研究グループは、標的とする細胞を特異的に除去する合成タンパク質をつくるために、死細胞の貪食に使用されるProtein Sを改変した。Protein Sのホスファチジルセリンを認識する領域(Glaドメイン)を、標的細胞に特異的な抗原を認識するドメインに置換することで、標的細胞を認識し、除去できるのではないかと考えた。
この新しい改変タンパク質は、「クランチ(Crunch:Connector for Removal of Unwanted Cell Habitat)」と命名された。クランチは、標的細胞に結合する「標的認識部位」、構造安定性を高める「EGF様ドメイン」、貪食受容体(MerTKなど)に結合する「SHBG様ドメイン」の3つの機能モジュールから構成され、不要な細胞と貪食細胞を橋渡しするとともに、貪食シグナルを活性化する役割を果たす。
マウスモデルでメラノーマ細胞の増殖抑制に成功
メラノーマ(悪性黒色腫)をマウスに移植し、7日後にメラノーマに発現するTYRP1に対するscFv(scTYRP1)を付加したクランチをマウスに投与したところ、メラノーマ細胞の増殖が顕著に抑制された。コントロールであるGFPに対するscFv(scGFP)を用いても同様の効果は得られなかったことから、scTYRP1がメラノーマ細胞だけを特異的に除去することが示された。
SLEモデルマウスでB細胞を除去、症状改善を確認
次に、全身性エリテマトーデス(SLE)タイプの自己免疫疾患を引き起こすFas(細胞死受容体)の欠損マウスにおいて、CD19に対するscFv(scCD19)を用いたクランチがB細胞を除去し、病態が回復するかを検討した。その結果、血液中のB細胞数の顕著な減少が確認された。また、腎臓に浸潤した白血球の数および抗体の沈着も減少し、尿中UCP ratioの正常化がみられた。血液中の抗DNA抗体や抗核抗体の濃度も低下しており、クランチによる治療効果が認められた。
不要細胞を除去する新たな疾患治療プラットフォームの確立に期待
今後、クランチを用いることで、臓器に存在する特定の細胞を除去したり、神経細胞などの大きな細胞の特定のコンパートメント(シナプスなど)を除去することで標的細胞やコンパートメントの機能を調べることが可能になると考えられる。また、クランチの体内での半減期を延ばすなどの技術開発により、さまざまなヒト疾患への応用が期待される。さらに、既存の抗体やCAR-Tの標的認識部位、あるいは臨床試験で十分な効果が得られなかった抗体やCAR-Tの標的タンパク質に対する認識部位をクランチに搭載することで、不要な細胞の除去を目的とした新たな治療戦略の提示にもつながる可能性がある。
「現在、クランチの実用化に向けて、元楽天メディカル共同CEOの虎石貴氏と共同でバイオテックスタートアップを設立中だ」と、研究グループは述べている。(QLifePro編集部)